Notícias
INFORME N.16 REDE CORONA-ÔMICA.BR-MCTI
Rede Corona-ômica.BR-MCTI informa possível nova variante da COVID-19 no interior de SP com mutação também encontrada na variante indiana

A Rede Vírus MCTI informa que a Rede Corona-Ômica.BR-MCTI, através do Instituto de Biotecnologia (IBTEC), Instituto de Biociências - UNESP Botucatu, do Instituto de Biociências, Letras e Ciências Exatas (IBILCE) - UNESP, São José do Rio Preto, do Laboratório de Pesquisa em Virologia da Faculdade de Medicina de São José do Rio Preto - FAMERP- e da Faculdade de Zootecnia e Engenharia de Alimentos da USP Pirassununga (FZEA-USP), reporta a identificação de uma possível nova linhagem, relacionada à B.1.1.28, em amostras da região de Araraquara (DRS-3), que apresenta a mutação L452R na proteína S dos SARS-CoV-2.
Informamos que, de 148 amostras dessa região que foram tipificadas pelo nosso grupo, 118 foram da variante P1, 17 como P2/N9, 4 como B1.1.7 e 9 como B.1.1.128 (nova variante). Isso ressalta a importância dessa nova variante que permanece em circulação onde predomina as variantes de preocupação (VOC).
A sequência de genoma completo de duas amostras coletadas em 24/03/2021 e 05/04/2021 na cidade de Porto Ferreira – SP, classificadas como B.1.1.28 pelo Pango lineages, apresentou a mutação L452R na proteína S. Devido à importância desta mutação, relacionada ao escape de anticorpos neutralizantes, estando a mesma presente nas variantes B.1.617 (Indiana), B.1.427 e B.1.429 (ambas da Califórnia), realizamos analises filogenéticas comparando com outras sequências da linhagem B.1.1.28, já depositadas no GISAID (Tabela 1). A análise revelou que as duas sequências de Porto Ferreira formam um grupo monofilético, com outras 17 sequências de cidades da mesma região (Figura 1), com alta sustentação de ramos. (Figura 2). Foram identificadas 13 mutações não-sinônimas e 5 sinônimas próprias do clado, sendo 4 na proteína S. As substituições que definem o clado encontram-se descritas na Tabela 2. A formação de um grupo monofilético com alto suporte de ramo, a presença de mutações características e a circulação em determinada região geográfica são fatores que sugerem que se trata de uma nova linhagem descendente de B.1.1.28.
Com o intuito de rastrear a frequência dessa variante na região, realizamos investigação de 64 amostras das cidades dessa região geográfica via sequenciamento Sanger da região RBD da proteína S, onde se encontra a mutação L452R. Foram identificadas outras 7 amostras contendo esta mutação, sendo 6 de Porto Ferreira – SP e uma de Descalvado – SP. O genoma completo dessas amostras está sendo sequenciado e será depositado no GISAID.
É importante destacar que a sequência mais antiga depositada desta possível nova linhagem data de Fevereiro de 2021 sugerindo um surgimento recente. Observa-se também a circulação em regiões que apresentam predomínio da linhagem P.1.
Tabela 1. Informações das sequências que compõe o clado. Em negrito as sequências depositadas por este grupo.
|
Numero de acesso GISAID |
Cidade |
Depositante |
|
EPI_ISL_1734841 |
Porto Ferreira |
Rede Corona-ômica (IBTEC-UNESP ) |
|
EPI_ISL_1734875 |
Porto Ferreira |
Rede Corona-ômica (IBTEC-UNESP ) |
|
EPI_ISL_1520132 |
Mococa |
Instituto Adolfo Lutz |
|
EPI_ISL_1520133 |
Mococa |
Instituto Adolfo Lutz |
|
EPI_ISL_1580269 |
Cesário Lange |
Rede de Vigilância Genômica (Vigenômica)/UNESP |
|
EPI_ISL_1625982 |
Itirapina |
Instituto Adolfo Lutz |
|
EPI_ISL_1625983 |
Araras |
Instituto Adolfo Lutz |
|
EPI_ISL_1795091 |
Tambaú |
Instituto Butantan |
|
EPI_ISL_1795092 |
Tambaú |
Instituto Butantan |
|
EPI_ISL_1795084 |
Tambaú |
Instituto Butantan |
|
EPI_ISL_1795085 |
Tambaú |
Instituto Butantan |
|
EPI_ISL_1795086 |
Tambaú |
Instituto Butantan |
|
EPI_ISL_1795413 |
Tambaú |
Instituto Butantan |
|
EPI_ISL_1795087 |
Santa Cruz Das Palmeiras |
Instituto Butantan |
|
EPI_ISL_1795088 |
Santa Cruz Das Palmeiras |
Instituto Butantan |
|
EPI_ISL_1795089 |
Santa Cruz Das Palmeiras |
Instituto Butantan |
|
EPI_ISL_1795090 |
Santa Cruz Das Palmeiras |
Instituto Butantan |
|
EPI_ISL_1795401 |
Rio Claro |
Instituto Butantan |
|
EPI_ISL_1795412 |
Sumaré |
Instituto Butantan |
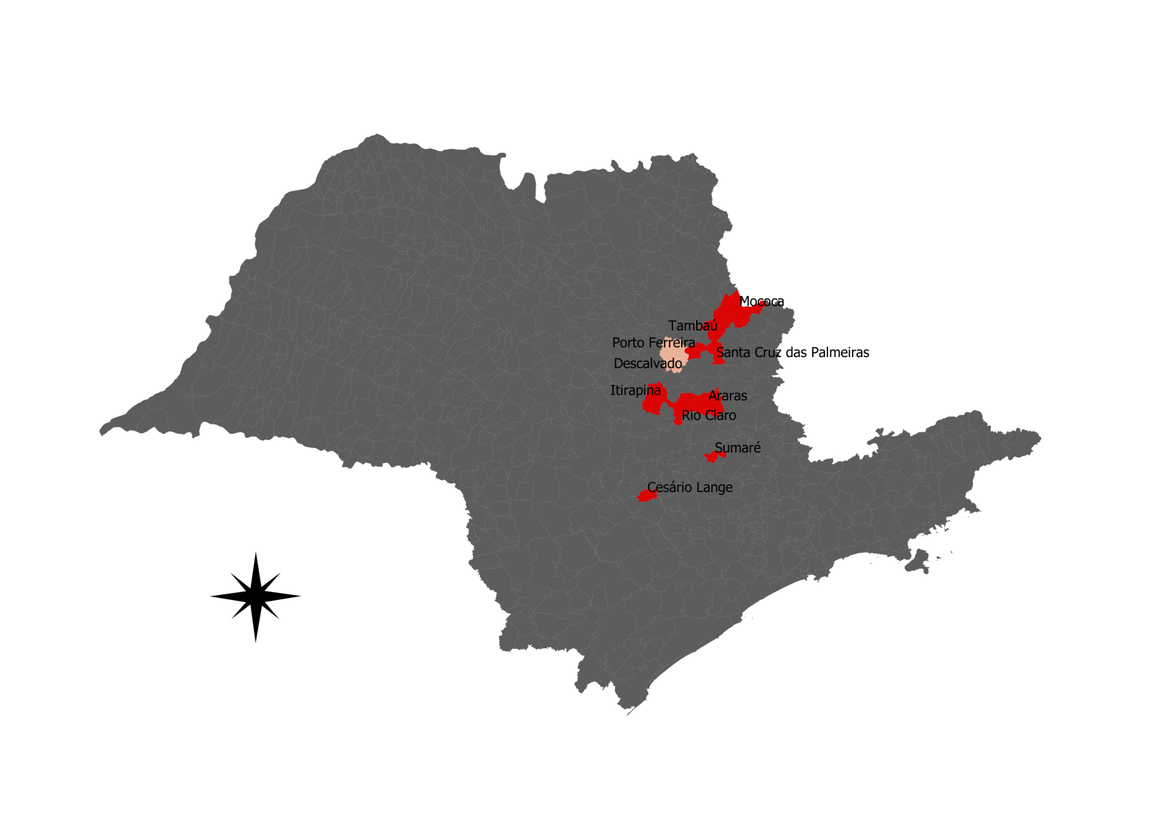
Figura 1: Mapa das cidades onde possível nova variante foi identificada. Vermelho – Cidades com sequências de genoma completo (19 amostras) . Bege – Cidade com sequenciamento da região RBD da proteína Spike (6 amostras)
Figura 1. A. Árvore filogenética de Maximum Likelihood reconstruída com base em um dataset de 337 sequências de genoma completo, incluindo B.1.1.28 e variantes descendentes (P.1, P.2, P.3). B. Detalhe do ramo da possível nova linhagem, apresentando a identificação das sequências e o suporte de ramo (aLRT). C. Detalhe apresentando as cidades onde as amostras de cada ramo foram coletadas.

Tabela 2. Mutações compartilhadas pelas sequências do clado (excluindo as já presentes na B.1.1.128 original) e a região genômica correspondente.
|
Região genômica |
Nucleotídeo |
Aminoácido |
|
A136G |
- |
|
|
ORF1a |
G1811A |
A516T |
|
ORF1a |
C3177T |
P971L |
|
ORF1a |
C9693T |
A3143V |
|
ORF1a |
T9867C |
L3201P |
|
ORF1a |
C11450A |
Q3729K |
|
ORF1a |
C12008T |
L3915F |
|
ORF1a |
C12880T |
- |
|
ORF1b |
A15932G |
Y822C |
|
ORF1b |
G18756T |
- |
|
ORF1b |
C20436T* |
- |
|
S |
G21987T |
G142V |
|
S |
C22079A* |
Q173K |
|
S |
T22917G |
L452R |
|
S |
A23720G** |
I720V |
|
ORF3a |
G25540A |
V50I |
|
A28271C |
- |
|
|
N |
C28932T |
A220V |
|
* Não estão presentes na sequência de Cesário Lange |
|
** Não está presente na sequência de Itirapina |
Informamos que envidaremos esforços para rastrearmos o surgimento dessa nova variante em amostras armazenadas da região e analisarmos dados epidemiológicos para determinação da sua possível origem para posterior publicação.
Devido à presença da mutação L452R, que é motivo de preocupação mundial, recomendamos às autoridades responsáveis que medidas sejam adotadas com urgência para evitar a dispersão desta possível nova linhagem para outras localidades.
Rede Corona-Ômica.BR-MCTI